Automatische Bilddatenanalyse in der RACOON-JIP Plattform
Die Möglichkeit zur föderierten Datenanalyse und Verarbeitung wird durch die JIP (DKTK Joint Imaging Plattform) in RACOON eingebracht. Die JIP wird lokal, innerhalb des Kliniknetzwerkes, installiert. Das modulare und container-basierte System ermöglicht die Verbindung als DICOM-Knoten mit den lokalen PACS-Systemen und bietet eine webbasierte Nutzerschnittstelle für die Bildbetrachtung und -annotation sowie die Durchführung komplexer Datenanalyse-Workflows.
Diese Workflows umfassen u.a. KI-Anwendungen zur Segmentierung und Berechnungen von Radiomics-Parametern, können aber auch komplette Werkzeuge wie beispielsweise MITK orchestrieren. Die JIP baut auf modernsten open source Komponenten auf, die sich als Industriestandard durchgesetzt haben. Die Integration von bestehenden Initiativen wie OpenMined und MONAI ermöglicht ein einfaches und sicheres föderiertes Training sowie die Anwendung von AI-Modellen für anspruchsvolle Datenanalyse-Workflows.
Im Rahmen von RACOON wird die Joint Imaging Platform als generische Plattform zur automatischen Bilddaten-Segmentierung und -Quantifizierung dienen. Zur Segmenterung wird die JIP hierzu Trainings- und Inferenzmöglichkeiten für nnU-Net, die aktuell führende Methode im Bereich der automatischen Segmentierung von medizinischen Bilddaten, liefern.
nnU-Net ist eine generische Segmentierungsmethode die in der Lage ist sich automatisch auf neue Aufgaben anzupassen und sich selbst zu konfigurieren, einschließlich Vorverarbeitung, Netzwerkarchitektur, Training und Nachbearbeitung. Die Methode zeichnet sich weiter dadurch aus dass sie besonders robust gegenüber variablen Eingabedaten ist und höchst performant in einer Vielzahl von verschiedenen Segmentierungsaufgaben lösen kann (siehe Grafik 1 und Grafik 2). Diese Eigenschaften machen die Methode ideal für den Einsatz in einem multizentrischen Szenario wie RACOON in dem robuste automatische Segmentierungen einer großen Anzahl von Datensätzen ein zentrales Element darstellen.
Zur Quantifizierung der mit nnU-Net segmentierten Strukturen und Pathologien stellt die JIP die Radiomics Methode zur Verfügung. Hierbei werden automatisch eine Vielzahl von Bild-basierten Merkmalen berechnet dann statistisch ausgewertet werden können, z.B. um den Schweregrad einer Erkrankung zu bestimmen oder einen möglichen Krankheitsverlauf vorherzusagen.
Die JIP wird lokal in den RACOON Nodes an jedem Standort alle empfangene Bilddaten der RACOON Kohorte verarbeiten. Die JIP stellt weiter entsprechende Workflows zum Training von nnU- Net sowie für die Radiomics Quantifizierung zur Verfügung. Ein besonders erwähnenswerter Aspekt hierbei ist, dass die JIP sogenanntes föderiertes Training von Methoden erlaubt. Hierdurch können die an den Standorten verteilt vorliegenden Daten, die aus Datenschutzrechtlichen Gründen nicht zentral gesammelt werden können, dennoch dezentral ausgewertet werden und die Ergebnisse fließen in einem optimierten KI Modell zusammen welches auf den Daten aller Standorte basiert.
Das der JIP zugrunde liegende System (Kaapana) ist öffentlich verfügbar, modular aufgebaut und basiert auf modernsten private-cloud Technologien die eine sichere und standardisierte Ausführung von Analysemethoden ermöglicht und es den einzelnen RACOON Standorten außerdem erlaubt, auf einfachem Wege eigene Analyse und Datenverarbeitungsmethoden an allen Standorten des RACOON Netzwerkes einzusetzen.

Semi-automatische und interaktive Segmentierung von Strukturen in der RACOON-SATORI Plattform
Die von Fraunhofer MEVIS entwickelten RACOON SATORI-Plattform kann auf allen RACOON-NODES genutzt werden und erlaubt eine effiziente Segmentierung von Lungenstrukturen und Pathologien auf CT-Daten (Grafik SATORI-1).
Die eingebundenen, trainierten neuronalen Netzwerke segmentieren automatisch anatomische Strukturen (Lungen, Lungenlappen, Bronchien und Gefäße) und Pathologien (Milchglastrübungen und Konsolidierungen). Ein speziell an die Anforderungen des RACOON-Konsortiums angepasster Workflow leitet die Benutzer:innen durch die Segmentierung eines Lungen-CTs. Der erste Schritt ist jeweils die Segmentierung der gesamten Lunge, welche die Basis für alle weiteren Strukturen stellt. BenutzerInnen können die durch KI automatisch erstellten Lungenmasken akzeptieren oder nachbessern. Für letzteres stellt SATORI interaktive Werkzeugen zur Verfügung. Besonders der Interpolationsmechanismus hat sich als ein effizientes Hilfsmittel herausgestellt, da die BenutzerInnen hiermit nicht mehr auf jeder von mehreren hundert CT-Schichten korrigieren müssen, sondern zum Beispiel nur auf jeder 10. Schicht.Nach der Segmentierung der gesamten Lunge folgen weitere anatomische Strukturen und alle im RACOON Konsortium definierten Lungenpathologien. In der SATORI Konfiguration ist hinterlegt, welche Pathologien sich ausschließlich innerhalb bzw. außerhalb der Lunge befinden. Wird eine Struktur, die nur innerhalb der Lunge existiert, über die Lungenränder hinaus gezeichnet, wird alles außerhalb der Lunge automatisch gelöscht. Diese Funktion spart NutzerInnen viel Zeit bei der Segmentierung.
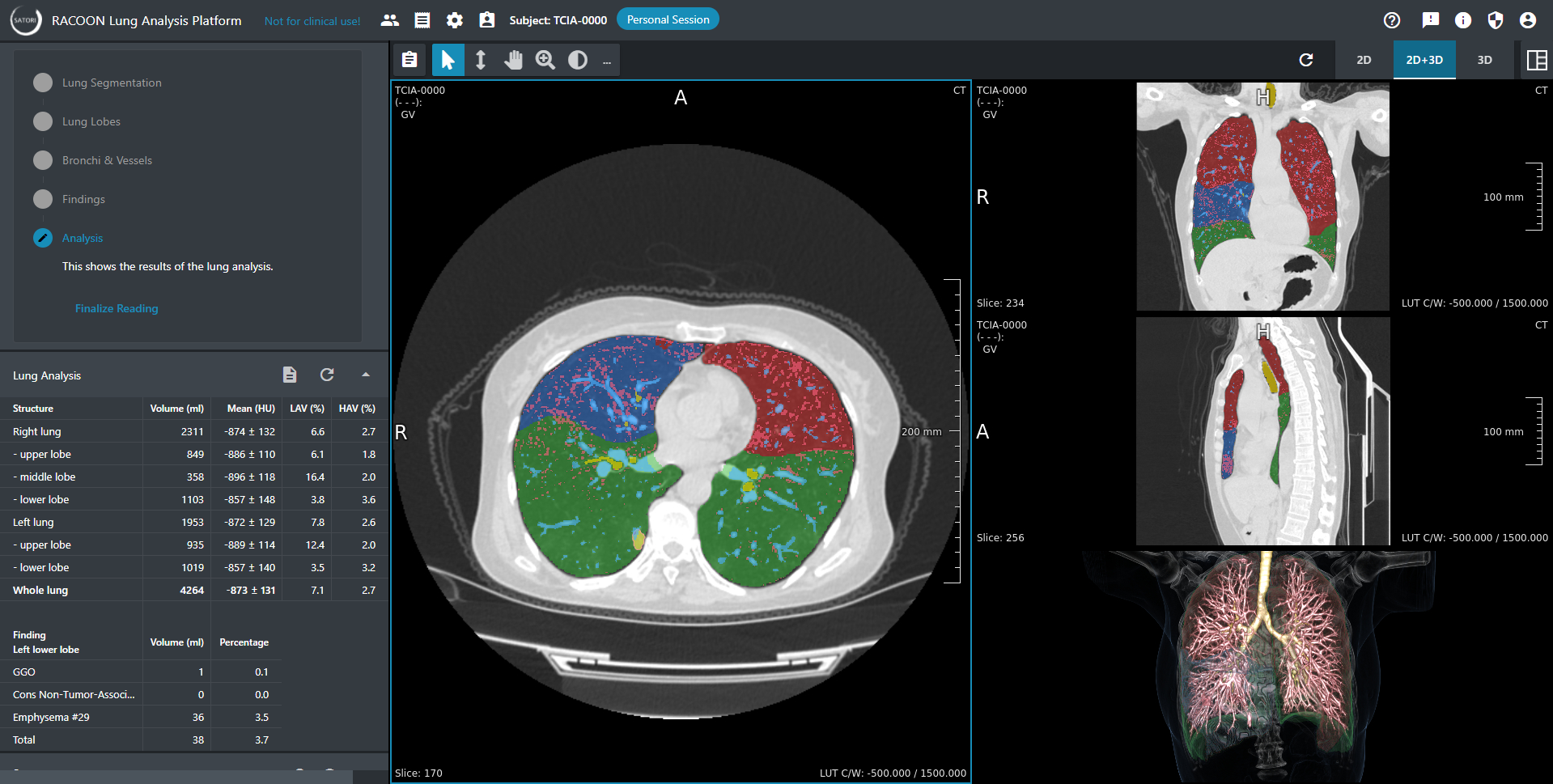
SATORI basiert auf einer Server-Client Architektur. Während das System in den Kliniken auf einem leistungsstarken Server mit GPU läuft, können BenutzerInnen über einen Browser auf ihren Desktop-PCs, Notebooks oder Tablets gemeinsam auf den Daten arbeiten. Der Import von CT-Daten und der Export der Segmentierungen erfolgt im DICOM-Standard und wurde in enger Zusammenarbeit mit den anderen technischen Partnern des RACOON Projektes optimiert.
SATORI ist hochgradig konfigurierbar und erweiterbar und kann dadurch leicht an individuelle Annotationsaufgaben angepasst werden. BenutzerInnen können zwischen verschiedenen Aufteilungen des Darstellungsbereichs (Hängungen) und Layouts wählen sowie Strukturen ein- und ausblenden. SATORI ermöglicht den Export von annotierten Screenshots der 2D-Ansichten sowie der 3D Visualisierung (Grafik SATORI-2). Ein Report fasst die Ergebnisse der quantitativen Analyse zusammen.
Dashboard & Qualitätsmanagement
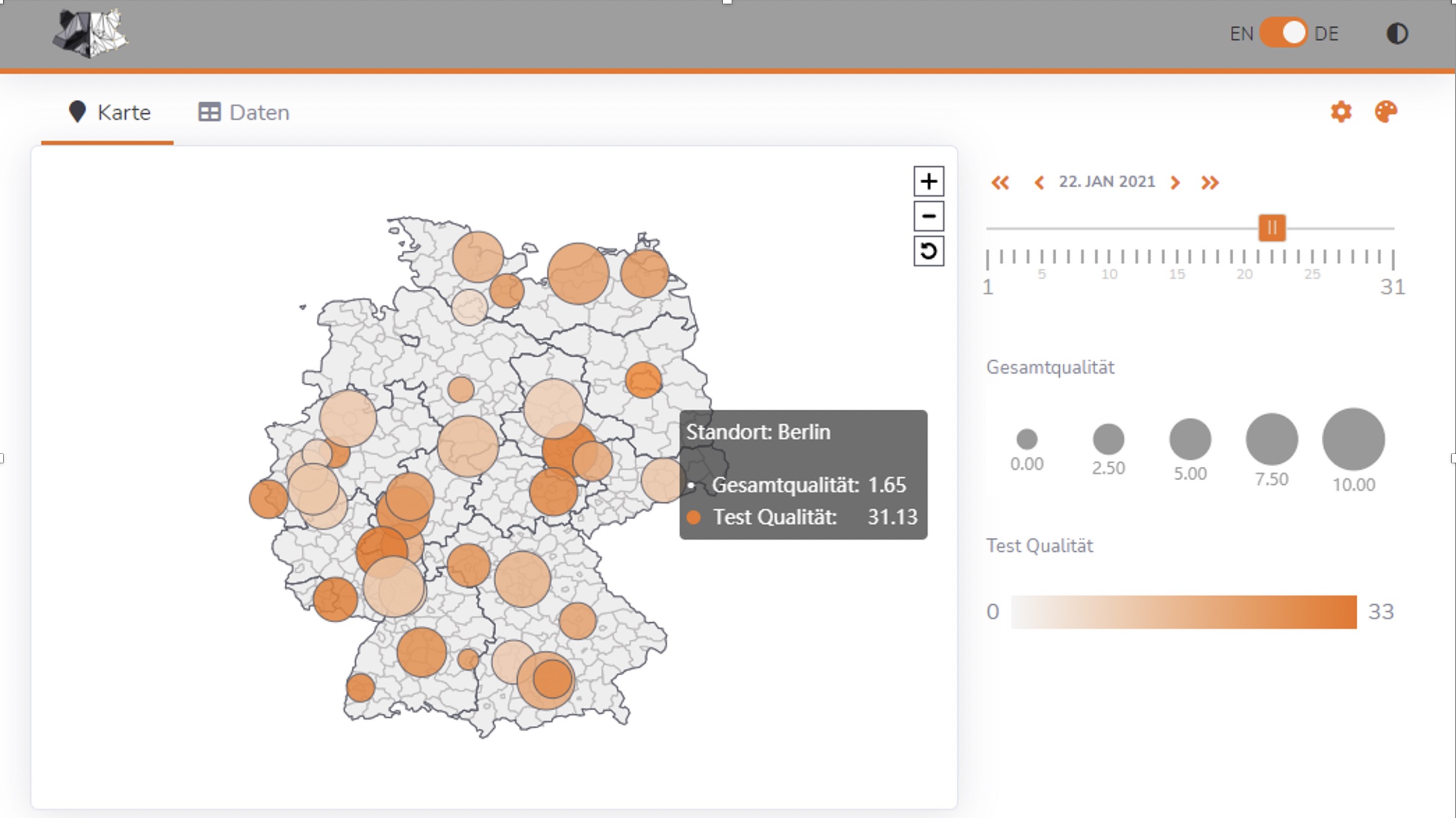
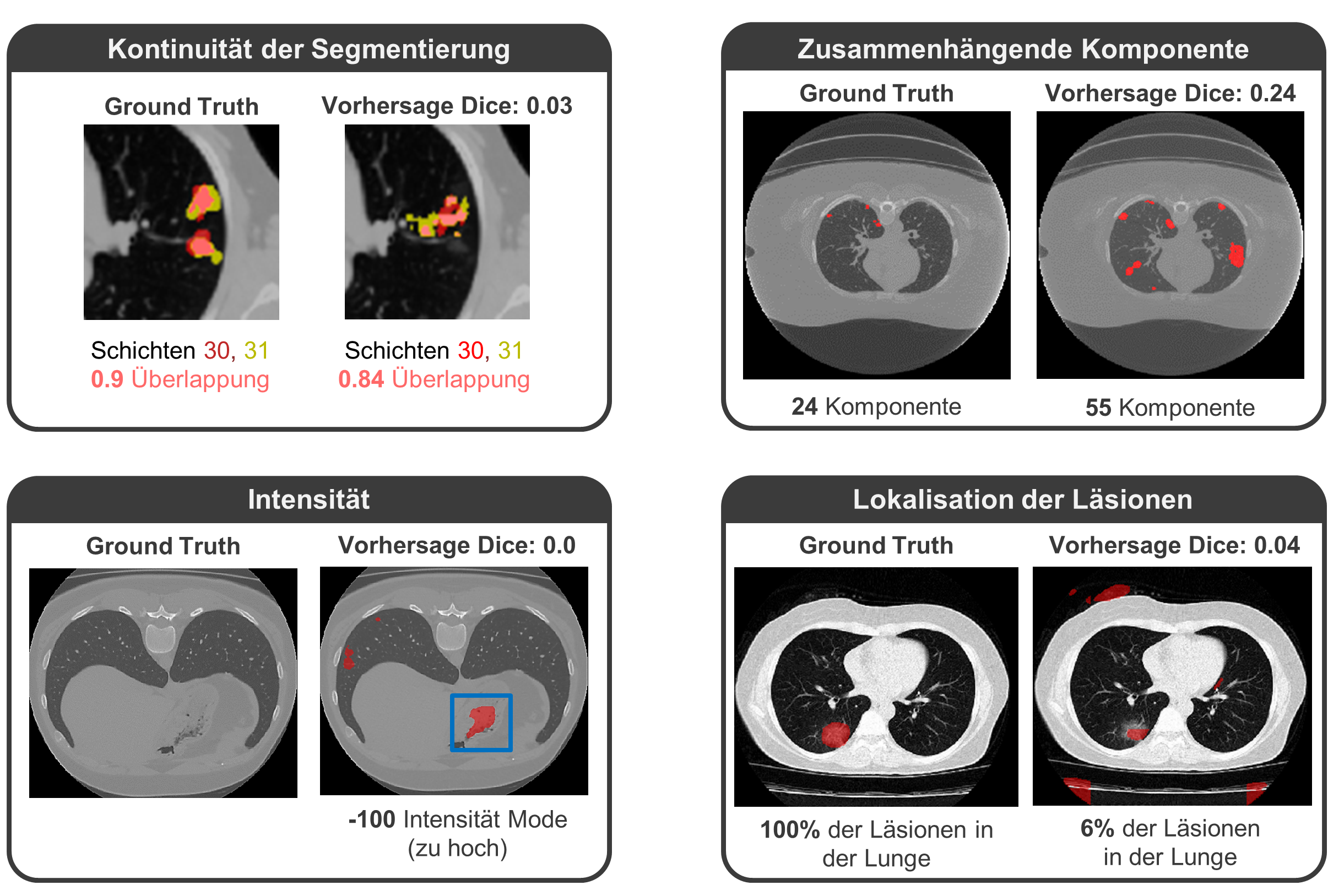
Die TU Darmstadt arbeitet im Rahmen des RACOON Projekts an der Entwicklung eines Verfahrens zur automatischen Qualitätssicherung, sowie der Entwicklung eines Visualisierungs-Dashboards zur Anzeige und Analyse von Statistiken. Um die Integrität der RACOON-Daten zu schützen, werden alle Metriken lokal in den einzelnen Standorten berechnet. Aggregierte Werte werden an den zentralen Knoten weitergeleitet und in der Visualisierungs-Dashboards angezeigt, die als JIP-Erweiterung implementiert ist.
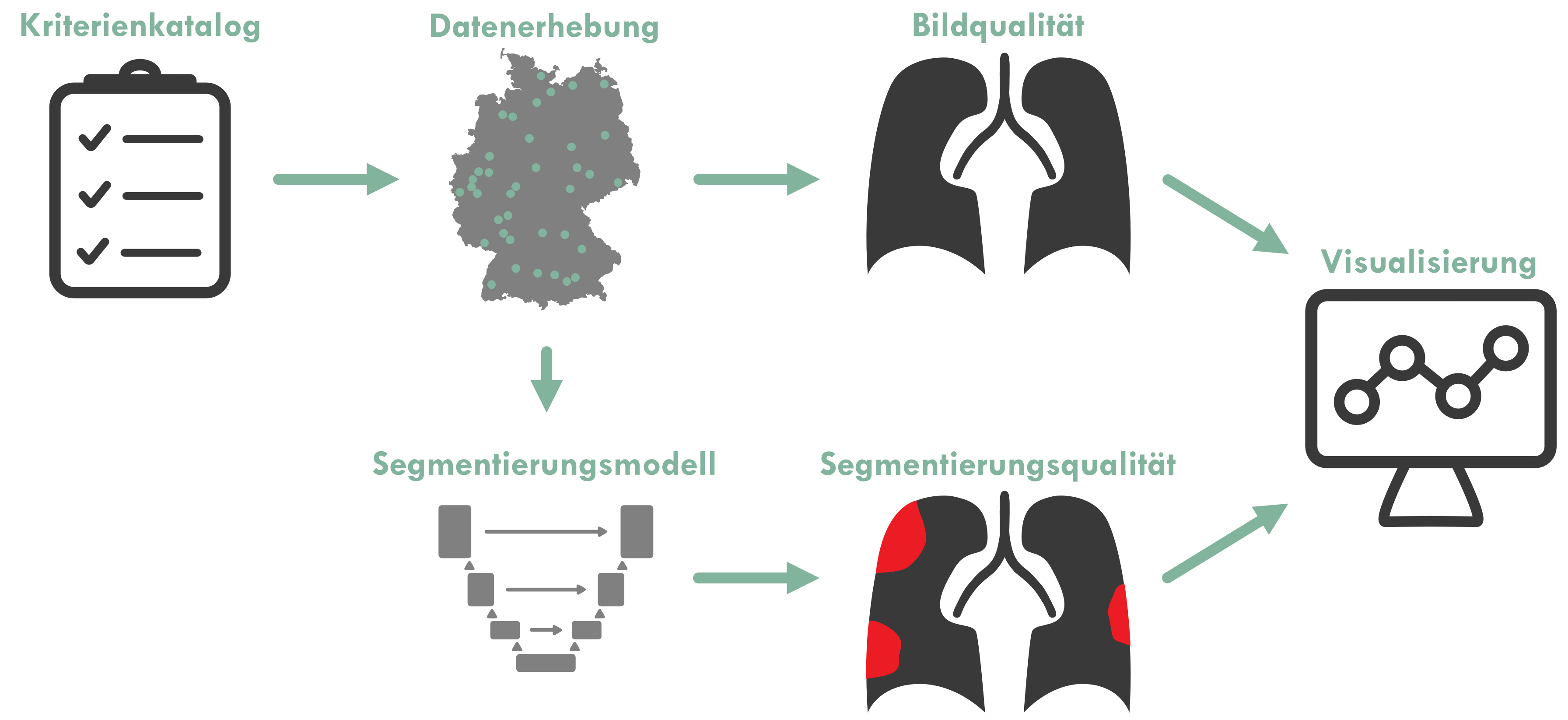
Die Qualitätssicherung dient in erster Linie dazu, die im Projekt verarbeiteten Bilddaten und Segmentierungsmasken automatisch zu evaluieren. Besonders für Segmentierungen, die mithilfe von KI-Modellen erzeugt wurden, ist das ein wichtiger Schritt. Die Qualitätssicherung kann in diesen Fällen sicherstellen, dass segmentierte Objekte in sich konsistent sind oder sie anhand von Größe und Kontinuität bewerten.